Wir leben in einer Informationsgesellschaft. Die Menge an Daten, die wir täglich erzeugen, speichern und verarbeiten, wächst exponentiell. Herkömmliche Speichertechnologie braucht Platz, ist fragil und hat einen gigantischen ökologischen Fußabdruck. DNA-Storage soll die Probleme lösen. Doch wie schnell geht das?
Write Once. Read Never?
70% aller Daten sind in Archiven gespeichert. Diese Daten werden selten bis nie wieder gelesen. Archivdaten werden auf speziellen, für die Langzeitarchivierung geeigneten Medien gespeichert. In den meisten Fällen sind das Tapes, manchmal auch optische Medien wie BlueRay oder LaserDisc. Doch auch bei diesen Medien spielt Haltbarkeit eine Rolle. Für Tapes geben Hersteller die Haltbarkeit mit 30 Jahren an. Wesentlich kritischer ist allerdings, wie lange es Hard- und Software gibt, mit der sich die archivierten Daten wieder auslesen lassen.
Die größten Datenarchive entstehen im wissenschaftlichen Umfeld. Aufzeichnungen von Klimadaten, Aufnahmen von Raumsonden, Ergebnisse aus Messreihen unzähliger Forschungsprojekte oder medizinische Daten sind nur einige Beispiele für Archive weltweit. Big Data und KI sind heute dankbare für all diese Daten. Technologie und Wissenschaft schreiten beständig voran und können heute Erkenntnisse aus Daten gewinnen, die Wissenschaftler vor ein paar Jahren noch als bedeutungslos oder unrichtig empfanden. So ignorierten z. B. Wissenschaftler in den 1970er Jahren die auf Weltraumflügen gesammelten, viel zu niedrigen Messwerte zu Ozondaten. In den 1980er Jahren wiesen britische Wissenschaftler auf eine gefährliche Ausdünnung der Ozonschicht hin. Mit den ursprünglich als fehlerhaft eingestuften Daten konnte das Ozonloch bewiesen werden.
Selbstverständlich ist das nur möglich, wenn sich die Daten noch auslesen lassen. Im Zuge neuer Theorien über die Entstehung von Leben suchte ein Wissenschaftler einen Nachweis von Lehm auf dem Mars. Hinweise gab es in den von der Viking-Mars-Sonde gesammelten Daten. Er hatte nicht so viel Glück wie die Klimaforscher. Es kostete ihn großen Aufwand und viel Zeit, die Daten auszuwerten, da niemand daran gedacht hatte, die Aufnahmen nach Ankunft auf der Erde entsprechend aufzubereiten.
Es gab zwar Kopien einiger alter Computerprogramme, mit denen die Rohdaten in Bilder umgewandelt wurden, aber die Quellcodes, die der Computer zum Ausführen der Programme benötigte, konnten nicht gefunden werden, und die Computer selbst existierten nicht mehr.
Eric Eliason, United States Geological Survey (Quelle: New York Times)
Die Faustregel lautet, auf Bändern oder optischen Medien archivierte Daten regelmässig umzukopieren. Die Empfehlung lautet alle fünf Jahre. Auch die NASA lernte dazu und begann im Sommer 1989, ihre bis dato angestauten 135.000 Bänder zu sortieren und für die Archivierung auszuwerten. Die ganze Geschichte gibt es im Archiv der New York Times zum Nachlesen.
Bis zur Unendlichkeit und noch viel weiter
Aktuell existieren schätzungsweise um die 33.000 Exabytes an Daten. Das sind 36.283.883.716.608 Megabyte oder 483 Milliarden Katzen – geht man davon aus, dass ein Bild mit dem neuen iPhone 14 ist 75 MB groß ist. In Cloudspeichern liegen derzeit nur ca. 550 Exabytes des gesamten Datenbestandes.
Die meisten Daten werden in den Branchen Gesundheit & Wissenschaftliche Forschung (36%), Finanzen (30%), Industrie (26%) und Medien (25%) erzeugt. In der Medizin sowie Forschung & Lehre sorgen vor allem die Bereiche Bildgebung, -Omics (vor allem die Genomik), personalisierte Medizin (Mikrobiota), Astrophysik, Teilchenphysik, Ökologie und Umweltwissenschaften für einen rasanten Anstieg der Datenmenge. Auch die Industrie mit dem Internet der Dinge (IoT), zunehmender Automatisierung und Zukunftstechnologie wie dem autonomen Fahren sowie Medien mit virtual und augmented Reality (VR/AR), Videostreaming, Online-Gaming und immer größerer Verbreitung der sozialen Netzwerke erzeugen mehr und mehr Daten.
IDC geht davon aus, dass sich die Datenmenge bis 2025 mehr als verfünffachen wird. Das bringt weitere Probleme mit sich. Bereits jetzt nimmt die Fläche der Rechenzentren weltweit den Platz von zwei europäischen Metropolen wie Paris in Anspruch. Der Ökofußabdruck entspricht dem der kompletten, weltweiten zivilen Luftfahrt. Mit 150 TWh pro Jahr verbrauchen diese Rechenzentren den Strom von 30 Atomkraftwerken. Und die Welt steht erst am Anfang einer wirklich umfassenden Digitalisierung. Inzwischen geht man auch davon aus, dass aktuelle Technologie nicht ausreichen wird, den künftigen Speicherbedarf zu decken.
Wenn wir heute in der Lage sind, 30% der von uns erzeugten Informationen zu speichern, werden wir in zehn oder zwölf Jahren in der Lage sein, nur etwa 3% zu speichern.
Dr .Karin Strauss, Senior Principal Research Manager bei Microsoft Research
Der Mensch als Speichermedium?
Abhilfe sollen DNA-Speicher schaffen.

Erste Versuche mit diesem Medium gab es bereits 1988. Damals wandelte der Künstler Joe Davis die Pixelinformationen eines Bildes in eine 0-1-Sequenz um, kodierte die Information in ein 28 Basenpaare-langes DNA-Molekül kodiert und baute es in ein Escherichia-Coli-Bakterium ein. Mittlerweile arbeiten mehrere Universitäten und Institutionen an DNA-Speicherlösungen. Die bekanntesten Projekte sind das der Goldmann Group am Bioinformatics Institute (EBI) der Cambridge Universität, das DNA-Data-Storage-Research-Projekt des Imperial College in London, das DNA-Storage-Projekt des WYSS Institute an der Harvard Universität, das Projekt MOSLA deutscher Universitäten (Marburg, Gießen) und Institute (LOEWE) oder das Laboratoire de Biologie Computationnelle et Quantitative an der Sorbonne Paris. Forschern des Frontiers Science Center for Synthetic Biology and Key Laboratory of Systems Bioengineering an der Tianjin Universität in China gelang jüngst ein Durchbruch mit einem neuen System. Auch in der Industrie forscht man rege an der neuen Technologie mit – allen voran Microsoft in Kooperation mit dem Molecular Information Systems Lab der Universität in Washington. aber auch Hardware-Hersteller sind ganz vorn mit dabei, wie ein Blick auf die Mitgliederliste der DNA Storage Alliance verrät. Und auch die SNIA fragt: „Ist DNA Storage die Zukunft?“
In der Natur gibt es eine andere Art von Datenspeicher, der nicht vom Menschen geschaffen wurde und seit fast 4 Milliarden Jahren verbessert wird.
Pierre Crozet, Maître de Conférences an der Sorbonne und Co-founder & CTO von Biomemory auf der IT Press Tour 2022 in Paris
Vor allem die hohe Dichte, die lange Haltbarkeit und die geringen Wartungskosten machen DNA-Storage so begehrenswert.
In einer menschlichen Zelle gibt es 6,4 Milliarden Nukleotidpaare, was einer Datenmenge von 700 MB entspricht. Der Mensch hat 3.900 Milliarden Zellen, was einer gespeicherten Datenmenge von 2,7 ZB gleichkommt. Um die aktuelle Menge an Daten zu speichern, würde man demnach 13 Menschen benötigen – mit Backup 26. Auch die Haltbarkeit von DNA ist beachtlich. Das älteste vollständige Genom stammt aus einem 1,6 Millionen Jahre alten Mammutzahn. Einmal als DNA gespeichert, sind Informationen leicht in unbegrenzter Menge zu reproduzieren. Aber keine Sorge, Menschen kommen aus diversen Gründen nicht als Speichermedium in Betracht.
DNA-Storage arbeitet mit einer synthetischen DNA. Der klassische Ansatz für DNA-Storage ist die Speicherung in Oligonukleotid-Pools. Oligos sind kurze Fragmente der DNA, die durch chemische Synthese gewonnen werden und maximal 200 Nukleotide beinhalten. Zu den Nachteilen gehören neben hohen Fehlerquoten vor allem die begrenzte Stückzahl und kostspielige Kopien.
Das Leben als Vorbild
Biomemory, ein Startup aus Paris, geht einen anderen Weg. Das Team um Pierre Crozet will das Potenzial der Natur nutzen, um die Beschränkungen der DNA-Speicherung zu beseitigen. Biomemory möchte einen biokompatiblen DNA-Storage entwickeln. Statt einzelsträngiger DNA-Moleküle, die viel weniger stabil sind als doppelsträngige DNA, entwickeln die Franzosen eine zirkuläre und replizierende doppelsträngige DNA, die mit sehr geringem Aufwand biologisch kopiert werden kann.
Dabei gibt es einige Herausforderungen zu bewältigen. Eine biokompatible DNA muss biosafe sein, das heisst, sie darf vom Wirtsorganismus nicht als genetische Information verwendet werden. Im Gegensatz zur chemischen Synthese sind nicht alle DNA-Sequenzen möglich. Mit dem Ansatz der biokompatiblen und biosafe DNA-Speichertechnologie sind längere Fragmente möglich und sie kann in vivo manipuliert werden. Das senkt die Kosten enorm.
Die französische Revolution in der DNA

Erste Erfolge verzeichnete das junge Team bereits mit einem spektakulären Proof of Concept. Zusammen mit den Archives Nationales, dem Programme Mémoire du Monde de l’UNESCO, der Sorbonne Université und CRNS sowie Partner aus der Industrie wie Twist Bioscience und Imagene AI konservierte Biomemory sowohl die Déclaration des Droits de l’Homme et du Citoyen von 1789) als auch die Déclaration des Droits de la Femme et de la Citoyenne von 1791 auf DNA-Basis.
Die Datei wurde zunächst kodiert und auf der DNA gespeichert und verkapselt. In mehreren Lesungen wurden die Daten mit 100%ige Genauigkeit wiedergegeben. Eine einzige Kapsel enthält mehr als 100 Milliarden Kopien der Datei auf DNA.
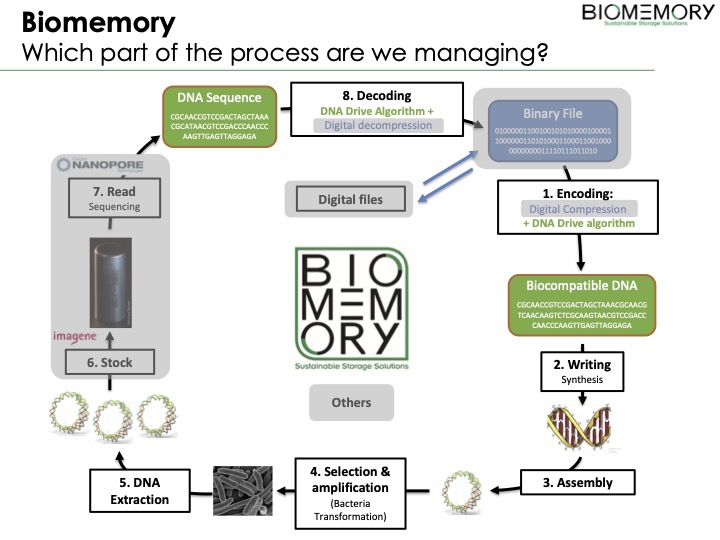
Der ganze Prozess des Kodierens und Entschlüsselns ist komplex. Und aktuell würde man im Rechnzentrum neben einem Labor auch einen Biologen und andere Wissenschaftler beschäftigen müssen. Allerdings gibt es erste Anwendungsfälle für die Technologie. Seit diesem Jahr wird DNA für das Backup von Kryptowährungen evaluiert. Ein solches Backup muss extremen Bedingungen standhalten und sehr langlebig sein. Ab 2024 soll die Technologie für die Rückverfolgbarkeit von Dokumenten, dazu gehört auch Geld, und Authentifizierungslösungen, z. B. fälschungssichere Pässe, eingesetzt werden. Gespräche mit einem weltweit führenden Hersteller von Sicherheitstinten für Banknoten und Ausweise laufen bereits. Ab 2030 soll der biokompatible DNA-Storage dann auch als Archivierungs- und Sicherungslösungen in Rechenzentren verfügbar sein.

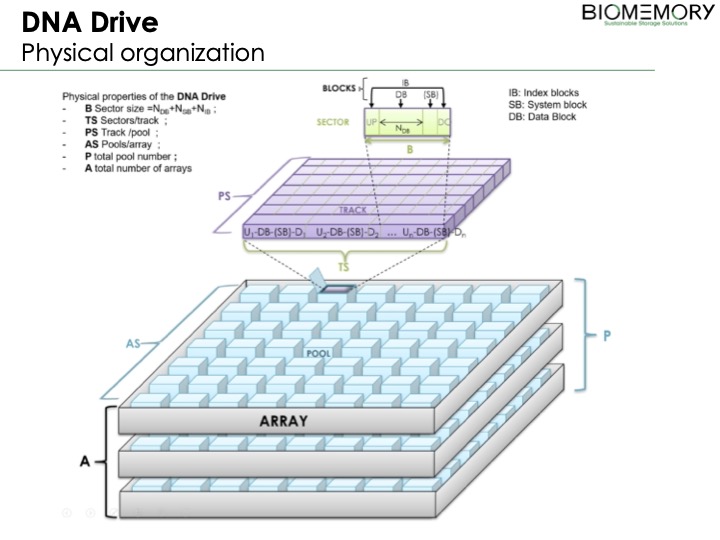
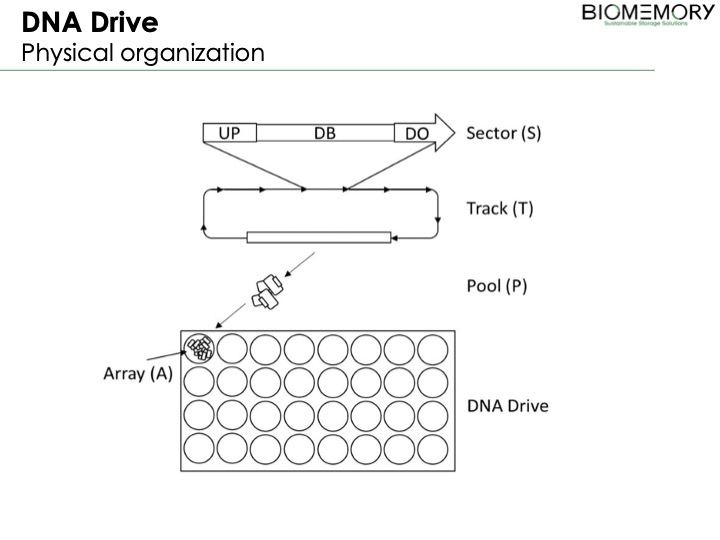
Wir sprachen mit den Gründern von Biomemory, CTO Pierre Crozet und CEO Erfane Arwani, auf der IT Press Tour in Paris.




